Resources:
将CRISPR编辑与细胞表型相关联
以单细胞分辨率将CRISPR编辑与基因表达表型直接关联。
获得更深入的见解
通过单细胞全转录组或靶向基因表达获得全面的见解,而不仅仅是细胞状态。
扩展您的CRISPR筛选
分析成千上万个细胞中数百个基因的遗传扰动效应。
缩短分析时间
与细胞存活分析所需的长时间培养相比,更快获得基因表达结果。
单细胞分辨率
逐个细胞地大规模分析疾病通路和扰动效应。
简化数据分析
通过易用的软件来探索扰动后的基因表达谱。
探索您的应用
可扩展的功能基因组学
揭示分化和发育的驱动因素
了解疾病生物学的机制
揭示整个基因组对疾病的遗传贡献
久经考验的结果
久经考验的结果
文献
搜索多篇采用Chromium单细胞CRISPR筛选的同行评审文献。
Combinatorial single-cell CRISPR screens by direct guide RNA capture and targeted sequencing
Combinatorial single-cell CRISPR screens by direct guide RNA capture and targeted sequencing
Nature Biotechnology. 2020, Replogle JM, et al.
Nature Biotechnology. 2020, Replogle JM, et al.
Genome-Wide CRISPR Screens in Primary Human T Cells Reveal Key Regulators of Immune Function
Genome-Wide CRISPR Screens in Primary Human T Cells Reveal Key Regulators of Immune Function
Cell. 2018, Eric Shifrut, et al.
Cell. 2018, Eric Shifrut, et al.
A Genome-Wide Framework for Mapping Gene Regulation via Cellular Genetic Screens
A Genome-Wide Framework for Mapping Gene Regulation via Cellular Genetic Screens
Cell. 2019, Molly Gasperini, et al.
Cell. 2019, Molly Gasperini, et al.
资源
查找有关Chromium单细胞CRISPR筛选的最新应用指南及其他文档。
Direct capture of guide RNAs enables scalable and combinatorial single cell CRISPR screens
Direct capture of guide RNAs enables scalable and combinatorial single cell CRISPR screens
Research Snapshot, 10x Genomics
Research Snapshot, 10x Genomics
Resolving the transcriptional events of life’s earliest stages with CRISPR and single cell RNA-sequencing
Resolving the transcriptional events of life’s earliest stages with CRISPR and single cell RNA-sequencing
Blog Article, 10x Genomics
Blog Article, 10x Genomics
完整的解决方案

Chromium单细胞基因表达试剂
使用我们的试剂盒,您可以通过3’mRNA、细胞表面蛋白、CRISPR扰动等来探索细胞多样性。

世界级的技术支持和客户支持
您可以通过电话或电子邮件联系我们的专家支持团队。
所需组件
单细胞CRISPR筛选可通过10x Genomics的下列组件来实现:
- Chromium Next GEM Single Cell 3' Kit v3.1
(1000269, 4rxns | 1000268, 16rxns)
- 3’ Feature Barcode Kit
(1000276, 16rxns)
- Chromium Next GEM Chip G Single Cell Kit
(1000127, 16rxns | 1000120, 48 rxns)
- Dual Index Kit TT Set A
(1000215, 96 rxns) - Gene Expression Libraries
- Dual Index Kit NT Set A
(1000242, 96 rxns) - *CRISPR Feature Barcode Libraries
- Targeted Gene Expression
CRISPR向导载体
将sgRNA克隆到AddGene提供的以下任一种与Feature Barcode兼容的载体中,构建您自己的CRISPR文库:
表达单个向导的CRISPR载体
表达两个向导的CRISPR载体
工作流程
- 1
设计CRISPR文库
选择您想要进一步研究其功能的目标基因——您既可以选择数十个基因来深入研究,也可以选择构成多条通路的数百个基因来广泛筛选。然后,从多个免费的CRISPR向导RNA设计工具中选择一个来设计您的sgRNA,并订购,将其克隆到采用10x Genomics捕获序列的CRISPR载体中。
资源 - 2
制备样本
转导并刺激细胞 - 感染细胞,选择表达CRISPR向导的细胞,然后施加刺激物。收集细胞,开始探索成千上万个细胞。按照洗涤、计数和浓缩细胞的最佳做法,尽量减少细胞团块的存在,并回收高质量的单细胞悬液。
资源 - 3
构建你的10x文库
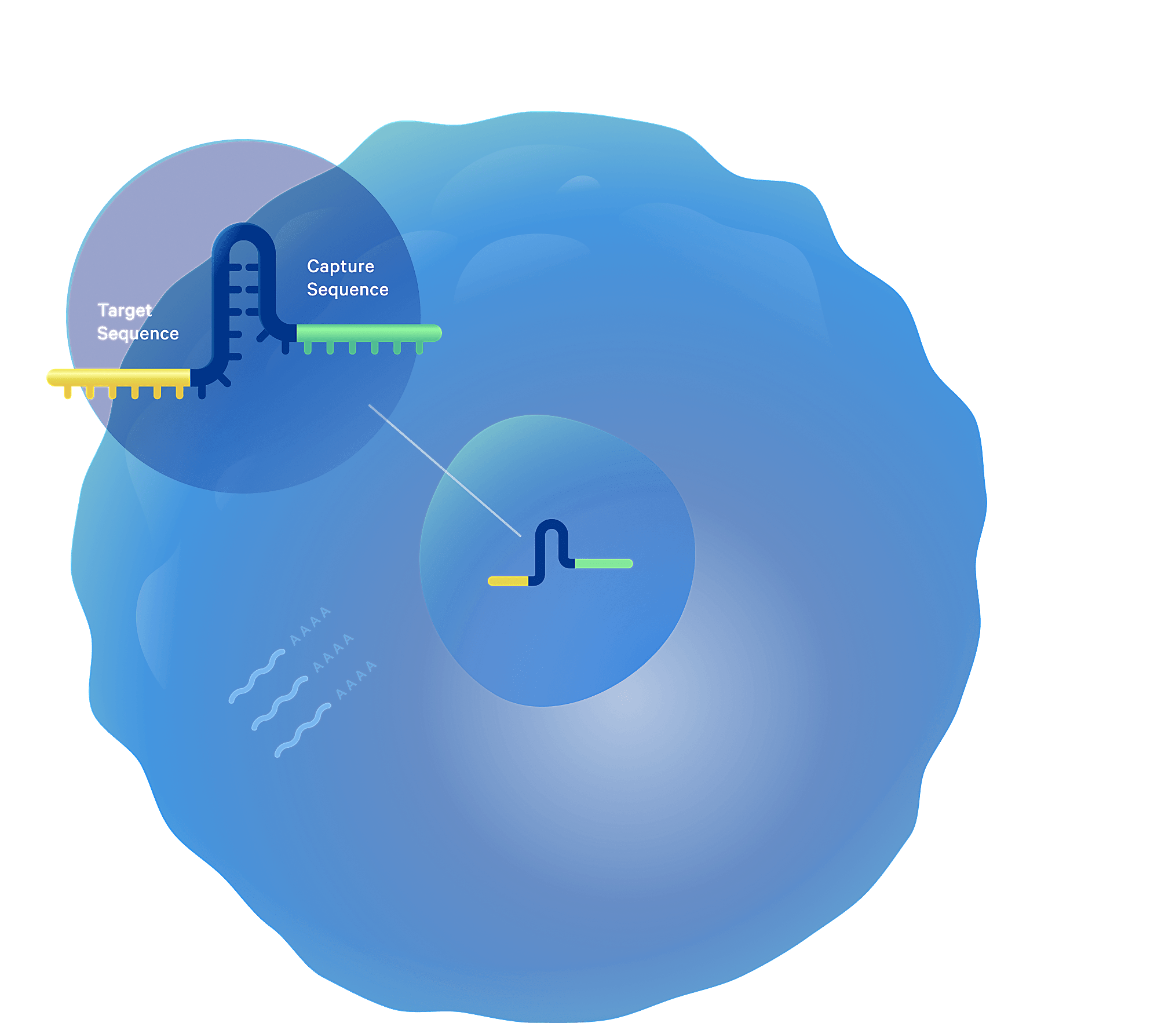
利用我们的试剂盒和Chromium Controller构建带有条形码的10x文库。Chromium Controller将每个带有10x条形码的凝胶珠的细胞封装在单个液滴中。在每个纳升级的液滴中,细胞经过逆转录,mRNA和CRISPR向导都生成cDNA,其中单个细胞的所有cDNA都带有共同的10x条形码。
单细胞CRISPR筛选是否与靶向基因表达兼容?
是的,您既可以探索全转录组的扰动效应,也可以使用靶向基因表达关注感兴趣的通路,通过测序成本的降低进一步扩大筛选规模。
资源 - 4
- 5
数据分析
Cell Ranger分析软件可自动为每个细胞分配向导,并直接评估向导对目标基因以及整个转录组的扰动效应。
分析流程输出
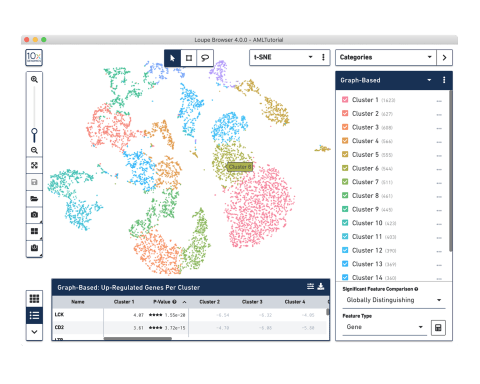
输出结果包括质控信息和文件,这些文件可轻松用于Loupe Browser可视化软件或第三方R工具及Python工具的进一步分析。
资源 - 6
数据可视化
使用我们的Loupe Browser可视化软件,您可以交互式地探索实验结果。研究目标基因的表达模式,分析各个向导靶点所产生的扰动表型,并开展细胞簇和样本之间的比较分析。
只有生物信息学家才能使用吗?
Loupe是点选式软件,任何人都可以轻松下载和使用。
资源
常见问题
来自10x Genomics的单细胞CRISPR筛选能够捕获每个细胞中的CRISPR sgRNA和细胞转录本并加上条形码,以混合向导文库的形式提供扰动和表型之间的直接关联。此外,分析读数提供了每个细胞的信息,能够分辨样本中的细胞簇和亚群。
CRISPR敲除、抑制和激活均经过了10x Genomics的结合Feature Barcode技术的Chromium单细胞基因表达解决方案的验证。
我们的兼容产品合作伙伴SigmaAldrich®*可以设计和制造与Feature Barcode兼容的定制慢病毒向导文库。采用Feature Barcode的CRISPR向导载体也可以在Addgene上找到,Weissman实验室存放了pBA900和pBA904载体。
SigmaAldrich® 10x Genomics定制CRISPR慢病毒产品
表达单个向导的CRISPR载体
表达两个向导的CRISPR载体
*Sigma-Aldrich is a trademark of Merck KGaA, Darmstadt, Germany or its affiliates. All other trademarks are the property of their respective owners. Detailed information on trademarks is available via publicly accessible resources.
基因编辑或功能基因组学核心实验室通常会提供资源,或者可以指导CRISPR向导的设计。Addgene也提供指导,帮助您选择最佳的CRISPR sgRNA设计工具。您也可以向MilliporeSigma提供目标基因列表,他们将为您订购的混合文库设计向导。
10x Genomics提供了两种类型的软件,可帮助您分析数据:Cell Ranger和Loupe Browser。Cell Ranger这款分析软件能够自动化生成每个细胞的转录图谱,并确定对基因表达水平的扰动效应。随后,您可以利用Loupe Browser可视化软件,通过聚类交互式探索整体的扰动差异,并比较不同扰动之间的差异基因表达。您可以从10x Genomics支持网站上免费下载Cell Ranger和Loupe Browser。