Resources:
クロマチンアクセシビリティを検出
ゲノムワイドに数千ものオープンクロマチン領域を細胞ごとに解析
シングルセル分解能
シングル核のエピゲノムプロファイルを測定し、集団平均値では見えにくい詳細な解析を実施
柔軟で高い拡張性
1チップあたり数百から数万個の核をプロファイル
効率的なデータ解析
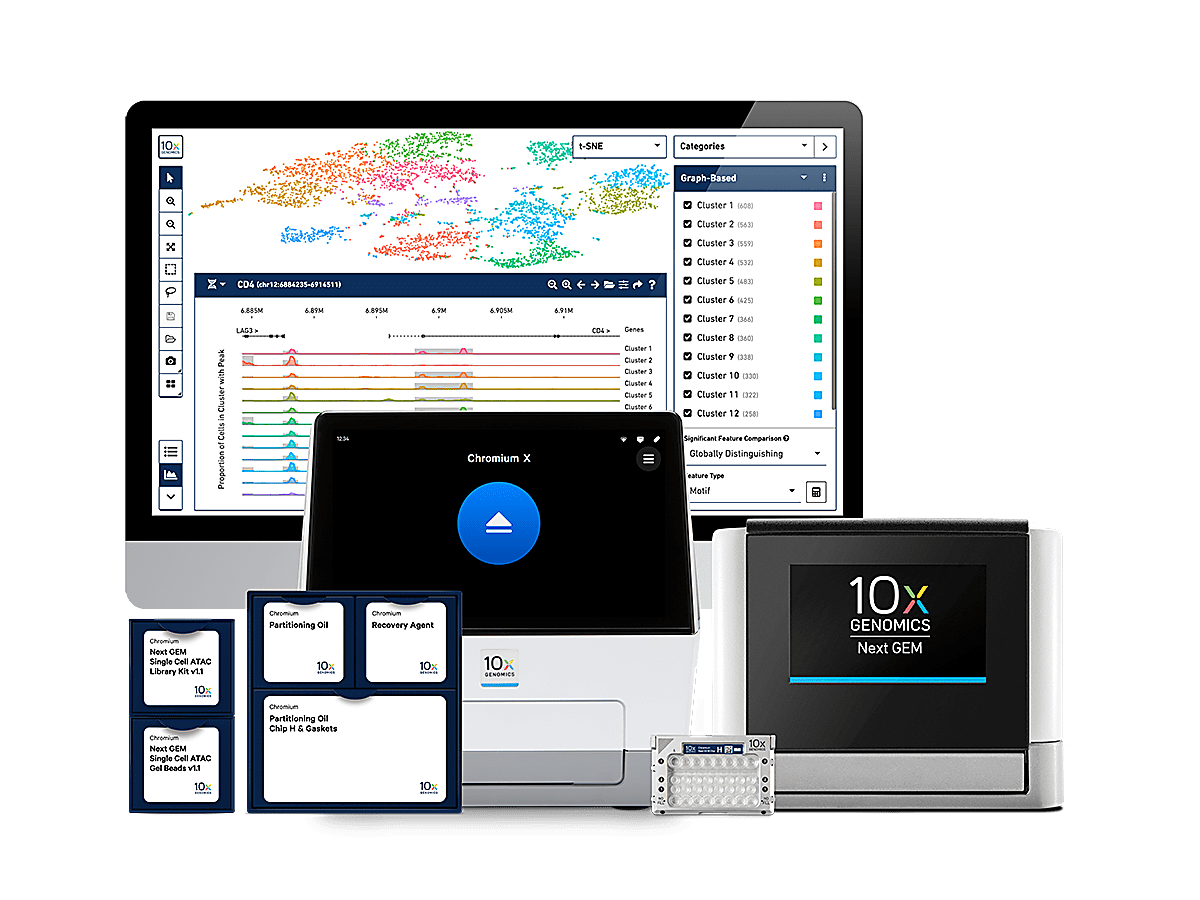
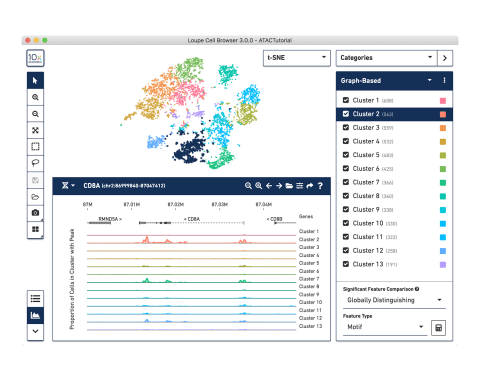
使いやすいソフトウェアでクロマチンアクセシビリティプロファイルを探索および視覚化
ラボ作業は1日
わずか1日でサンプルからシーケンス可能なライブラリーを作製
多様なサンプルに対応
細胞株、初代細胞、新鮮・冷凍組織サンプルからの検証済みプロトコール
さらなる可能性を探る
細胞の種類と状態を定義
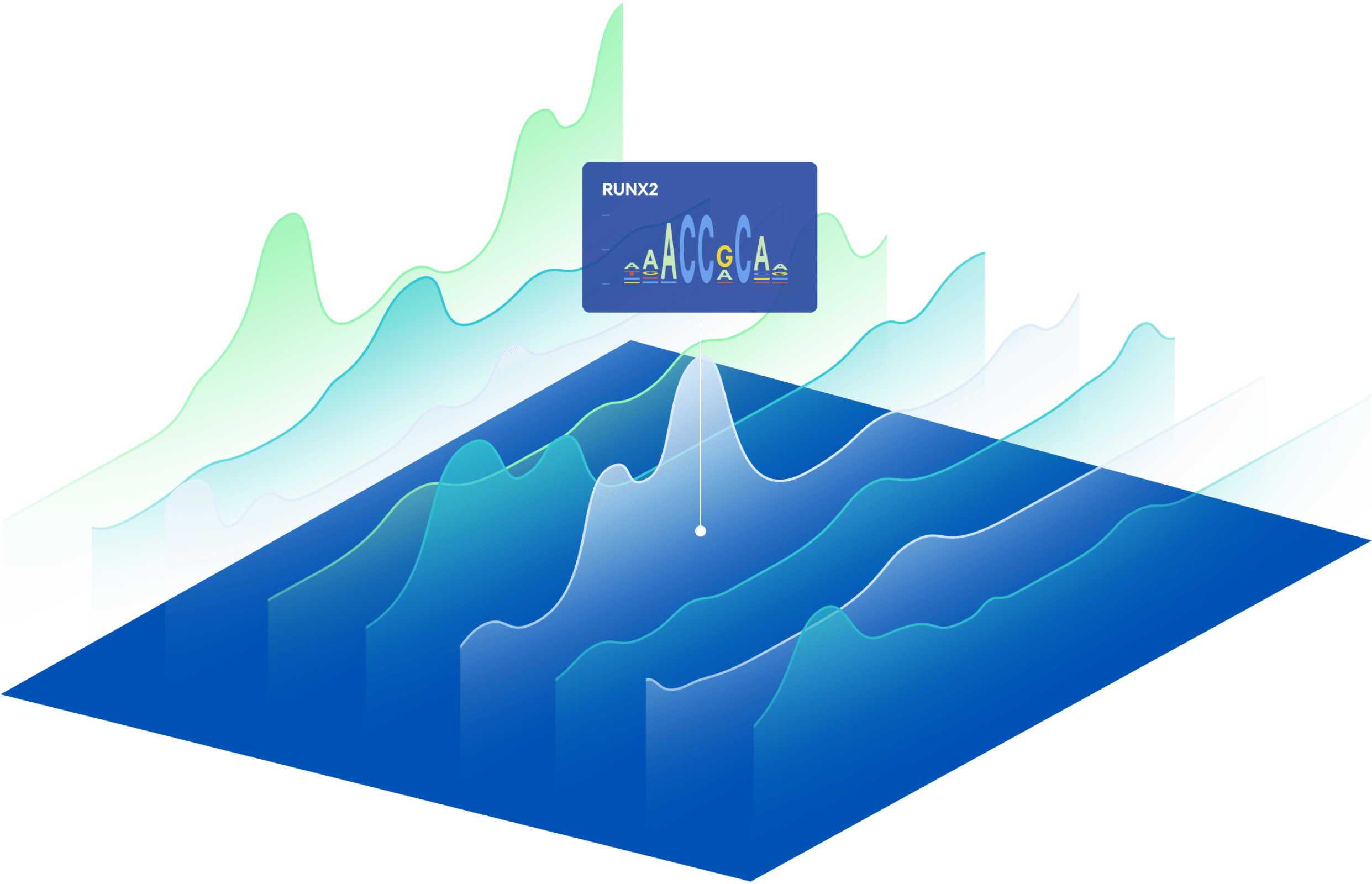
マスター制御因子の同定
プロモーターやエンハンサーなどのシス制御エレメントの発見
遺伝子制御ネットワークを特徴づけ
実績のある結果
実績のある結果
論文
数百にもわたる論文からChromiumシングルセルATACを使った結果を検索できます。
Single-cell multiomic analysis identifies regulatory programs in mixed-phenotype acute leukemia
Single-cell multiomic analysis identifies regulatory programs in mixed-phenotype acute leukemia
Nature Biotechnology, 2019, Jeffrey M Granja, et al.
Nature Biotechnology, 2019, Jeffrey M Granja, et al.
Massively parallel single-cell chromatin landscapes of human immune cell development and intratumoral T cell exhaustion
Massively parallel single-cell chromatin landscapes of human immune cell development and intratumoral T cell exhaustion
Nature Biotechnology, 2018, Ansuman T Satpathy, et al.
Nature Biotechnology, 2018, Ansuman T Satpathy, et al.
Hic1 Defines Quiescent Mesenchymal Progenitor Subpopulations with Distinct Functions and Fates in Skeletal Muscle Regeneration
Hic1 Defines Quiescent Mesenchymal Progenitor Subpopulations with Distinct Functions and Fates in Skeletal Muscle Regeneration
Cell Stem Cell. 2019, R Wilder Scott, et al.
Cell Stem Cell. 2019, R Wilder Scott, et al.
Rapid Generation of Somatic Mouse Mosaics with Locus-Specific, Stably Integrated Transgenic Elements
Rapid Generation of Somatic Mouse Mosaics with Locus-Specific, Stably Integrated Transgenic Elements
Cell. 2019, Gi Bum Kim, et al.
Cell. 2019, Gi Bum Kim, et al.
リソース
ChromiumシングルセルATACの最新アプリケーションノートや製品資料をご覧ください。
Deciphering Epigenetic Regulation with Single Cell ATAC-Seq
Deciphering Epigenetic Regulation with Single Cell ATAC-Seq
Application Note, 10x Genomics
Application Note, 10x Genomics
Single Cell ATAC-Seq for Characterization of Complex Biological Systems
Single Cell ATAC-Seq for Characterization of Complex Biological Systems
Application Note, 10x Genomics
Application Note, 10x Genomics
エンドツーエンドのソリューション

Chromium シングルセルATAC試薬
オープンクロマチン領域を検出するトランスポゼース酵素、NGSシーケンス可能なライブラリーの作製用試薬キット

グローバルな技術・カスタマーサポート
専門サポートチームがメールと電話で対応
ワークフロー
- 1
- 2
10xライブラリーの構築
試薬キットとChromium Controller装置を使用して、10xバーコードを付加したシングルセル ATACライブラリーを構築します。まず最初に、核をバルクでトランスポーズ処理します。次に、Chromium Controllerで10xバーコードがついたゲルビーズを加えて、それぞれの核を分画化します。ナノリットル単位で分画された液滴内部では、トランスポーズ処理したDNA断片を10xバーコードでラベル化します。1つの核から作成されたDNA断片には、共通の10xバーコードが付加されています。
リソース - 3
シーケンス
10xバーコードが付加されたライブラリーは、標準的なNGSショートリードシーケンサー(イルミナ)と互換性があります。数千個にもわたるそれぞれの細胞から、大量の転写プロファイリングを実施します。
リソース - 4
データ解析
Cell Ranger ATAC 解析ソフトウェアは、各細胞のオープンクロマチンプロファイルを生成し、類似したプロファイルを示す細胞のクラスターを同定します。さらにピークを検出し、複数のサンプルのデータを集約します。
解析パイプライン出力
出力にはQCの情報およびファイルが作成され、10xのLoupe視覚化ソフトウェア、サードパーティのR、Pythonツールで使用できます。
リソース - 5
データの視覚化
Loupe Browser 視覚化ソフトウェアを使用して、結果をインタラクティブに探索します。興味のある遺伝子の発現パターンを調べたり、細胞タイプを決定してクラスターとサンプル間の比較解析をしたりできます。
バイオインフォマティシャンが必要ですか?
Loupeは誰でも簡単にダウンロードして使用できるクリックベースのソフトウェアです。
リソース
よくある質問
オープンクロマチンのプロファイリングをシングルセルの分解能で実施できます。
はい、サードパーティ製のツールを使用することで可能です。詳細については、ウェビナーをご覧ください。
まず、核の懸濁液が必要です。新鮮な細胞、凍結保存された細胞、新鮮な組織、凍結組織から高品質な核の懸濁液を得ることができます。このアッセイはヒトとマウスのサンプルに最適化されていますが、他の生物種でも可能です。
バルクATAC-seqと細胞株由来のシングルセルATAC-seqの比較では、シングルセルATAC-seqはバルクATAC-seqで回収されたピークの90%以上を回収することが示されています。また、シングルセルATAC-seqは、細胞種類に特異的なレベルでクロマチンプロファイルを行う分解能を提供します。
ATAC-seqは特定の因子に結合している領域だけでなく、すべてのオープンクロマチン領域を捉えます。サンプル中のエピジェネティックな変化の探索には、ATAC-seqはバイアスのないアプローチです。結合モチーフ配列が既知のタンパク質の場合、付属のソフトウェアを使用することで細胞ごとにオープンクロマチン内のモチーフを特定できます。